Blog Más de cien líneas élite de arroz para América Latina y el Caribe serán adoptadas, procedentes de los programas de mejoramiento del arroz (Alianza-FLAR)

Por Nicolás Hoyos y Robert Andrade
Año tras año, cientos de líneas élite de arroz son desarrolladas por los programas de mejoramiento de la Alianza y FLAR, pero "¿cuánta diversidad tenemos?" y "¿cuál es la forma más eficiente de estudiarla?" son preguntas que siguen siendo relevantes.
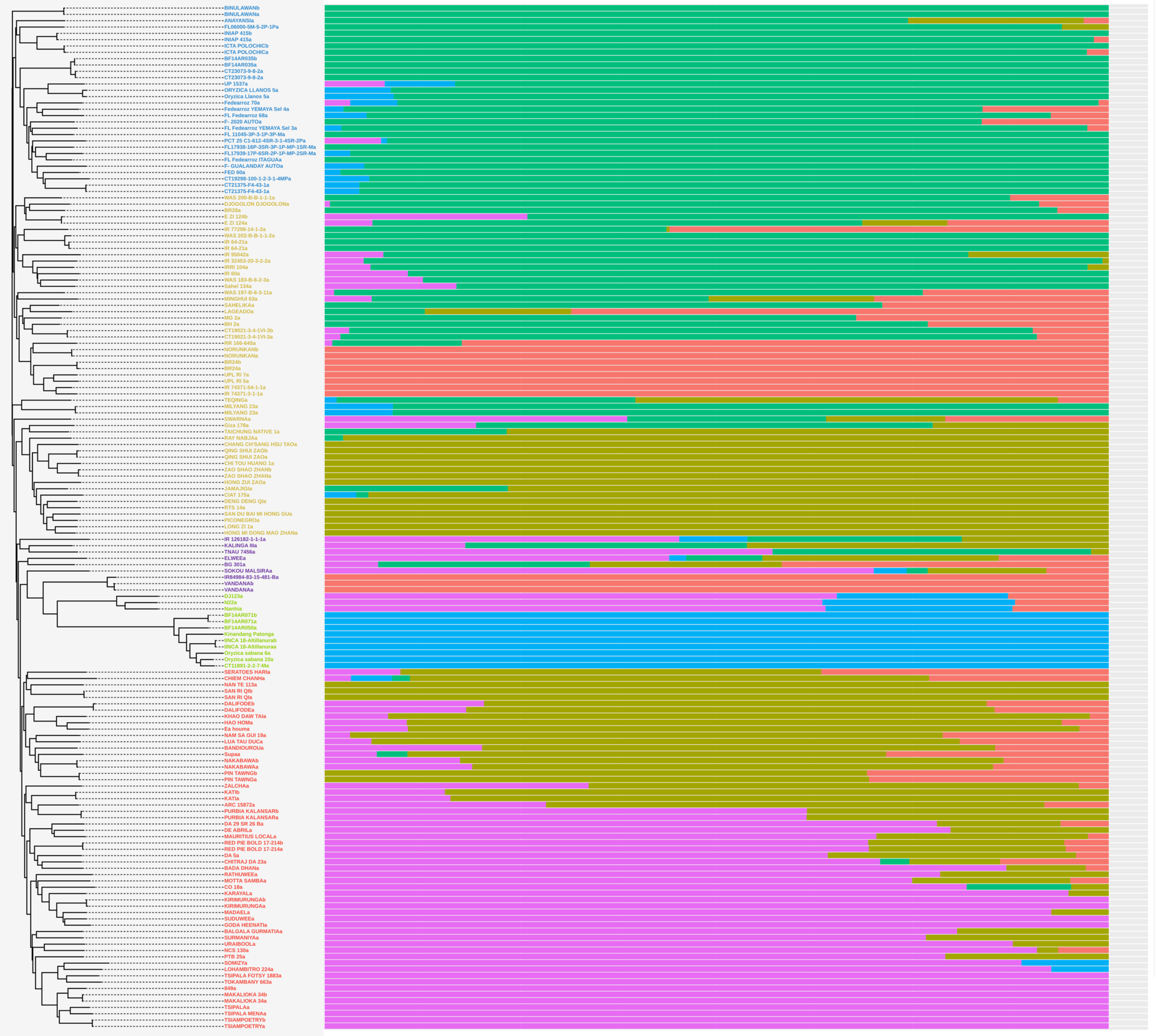
La genómica ofrece una enorme variedad de posibilidades tanto para la investigación como para la mejora de cultivos, la predicción de rasgos agrícolas relevantes, las interacciones genotipo-fenotipo y la mejora dirigida, por nombrar sólo algunas. Por este motivo, los programas de mejoramiento del arroz decidieron realizar la secuenciación completa de 137 variedades élite de arroz y llevar a cabo análisis de diversidad y genética de poblaciones. Determinamos que, debido al extenso historial de cruces dentro de las diferentes líneas, una estructura de árbol es inadecuada para mostrar las relaciones entre nuestras líneas, y el análisis de mezcla proporciona una imagen más precisa de su parentesco.
La metodología utilizada consistió en secuenciar con una profundidad media de 25x a más de 60x, pero nuestros resultados mostraron que la mayoría de las variantes ya estaban determinadas a 14x; nuestra primera recomendación es que, a menos que se vaya a estudiar el análisis estructural profundo o el número de copias de repeticiones, la profundidad de secuenciación objetivo no debería ser más de 20x, ya que los recursos se utilizarían de manera más eficiente en analizar más líneas en lugar de lograr una mayor resolución mediante una secuenciación más profunda.
Para aprovechar un contexto más amplio proporcionado por el vasto conocimiento disponible para la genómica del arroz, enriquecimos nuestro conjunto de datos con líneas seleccionadas del proyecto de 3.000 genomas (el proyecto de los 3.000 genomas de arroz), ya que proporciona información exhaustiva sobre toda la diversidad del arroz cultivado en el mundo. Al unir árboles vecinos, nos dimos cuenta de que las ramas resultantes eran muy inestables y cortas en longitud, probablemente debido a cruces recientes. También se observaron los individuos híbridos cuando se agruparon utilizando mezcla, lo que mostró mucha más estabilidad y fue consistente con y sin enriquecimiento con datos de 3.000 genomas.
Algunos de nuestros resultados más interesantes se encuentran dentro del análisis de diversidad con la estadística de diversidad pi y las curvas MAF. Comparamos la diversidad observada entre nuestro panel y la encontrada en el proyecto de 3.000 genomas, la cual es representativa de la diversidad global. Encontramos que para Indica, aunque la diversidad es menor, las líneas que analizamos tenían una estadística pi muy similar a la diversidad global; mientras que para las variedades genéticas Japonica y Aus fue notablemente menor. Esto implica que la inclusión de líneas de estos orígenes podría aumentar en gran medida la diversidad disponible.
Aunque nuestro estudio proporciona resultados esclarecedores, aún queda mucho trabajo por hacer. Buscamos realizar análisis de introgresión para identificar secciones del genoma que se han transferido entre grupos. Utilizar la caracterización fenotípica y realizar GWAS para buscar variantes relacionadas con rasgos relevantes para la agricultura. Utilizar los datos de adopción para calcular el incremento de producción generado por el programa de mejoramiento de arroz, teniendo en cuenta que estudios previos estiman que más de dos tercios de toda la superficie de arroz sembrada lo está con algún material relacionado con los programas de mejoramiento de arroz del CIAT y/o FLAR (Yamano et al., 2016) (Labarta et al., 2017). De igual forma, seguir agregando variantes a nuestra base de datos, para tener una mejor idea no solo de lo que hemos logrado, sino también del potencial futuro y las formas más efectivas de explotarlo. La cooperación entre mejoradores, genetistas, agricultores, productores de semillas, industria y economistas del desarrollo agrícola es primordial para hacer el mejor uso de los recursos genéticos. Por último, debemos fortalecer la producción de arroz en una región que durante décadas ha mantenido tasas de crecimiento en sus rendimientos promedio (Mishra, et al. 2022).
Referencias:
- El proyecto de los 3.000 genomas del arroz. (2014). GigaScience, 3(1). https://doi.org/10.1186/2047-217x-3-7
- Labarta, R., Andrade, R., Salazar, D. M., Rivera, T., Orrego, M., & Pinillos, J. (2017). Los impactos de la investigación colaborativa del CIAT. Centro Internacional de Agricultura Tropical (CIAT). Cali, CO. 12 p.
- Mishra, A. K., Pede, V. O., Arouna, A., Labarta, R., Andrade, R., Veettil, P. C., Bhandari, H., Laborte, A. G., Balie, J., & Bouman, B. (2022). Ayudando a alimentar el mundo con las innovaciones en arroz: adopción de la investigación de CGIAR e impacto socioeconómico en los agricultores. Seguridad Alimentaria Mundial, 33, 100628. https://doi.org/10.1016/j.gfs.2022.100628
- Yamano, T., Arouna, A., Labarta, R. A., Huelgas, Z. M., & Mohanty, S. (2016). Adopción e impactos de las tecnologías internacionales de investigación sobre el arroz. Seguridad Alimentaria Mundial, 8, 1-8. https://doi.org/10.1016/j.gfs.2016.01.002
